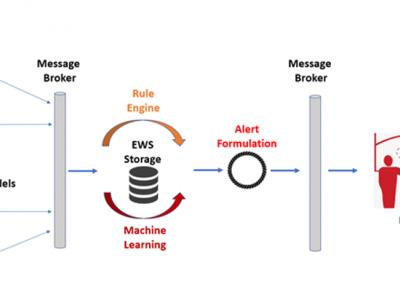
Contact
Provider(s):
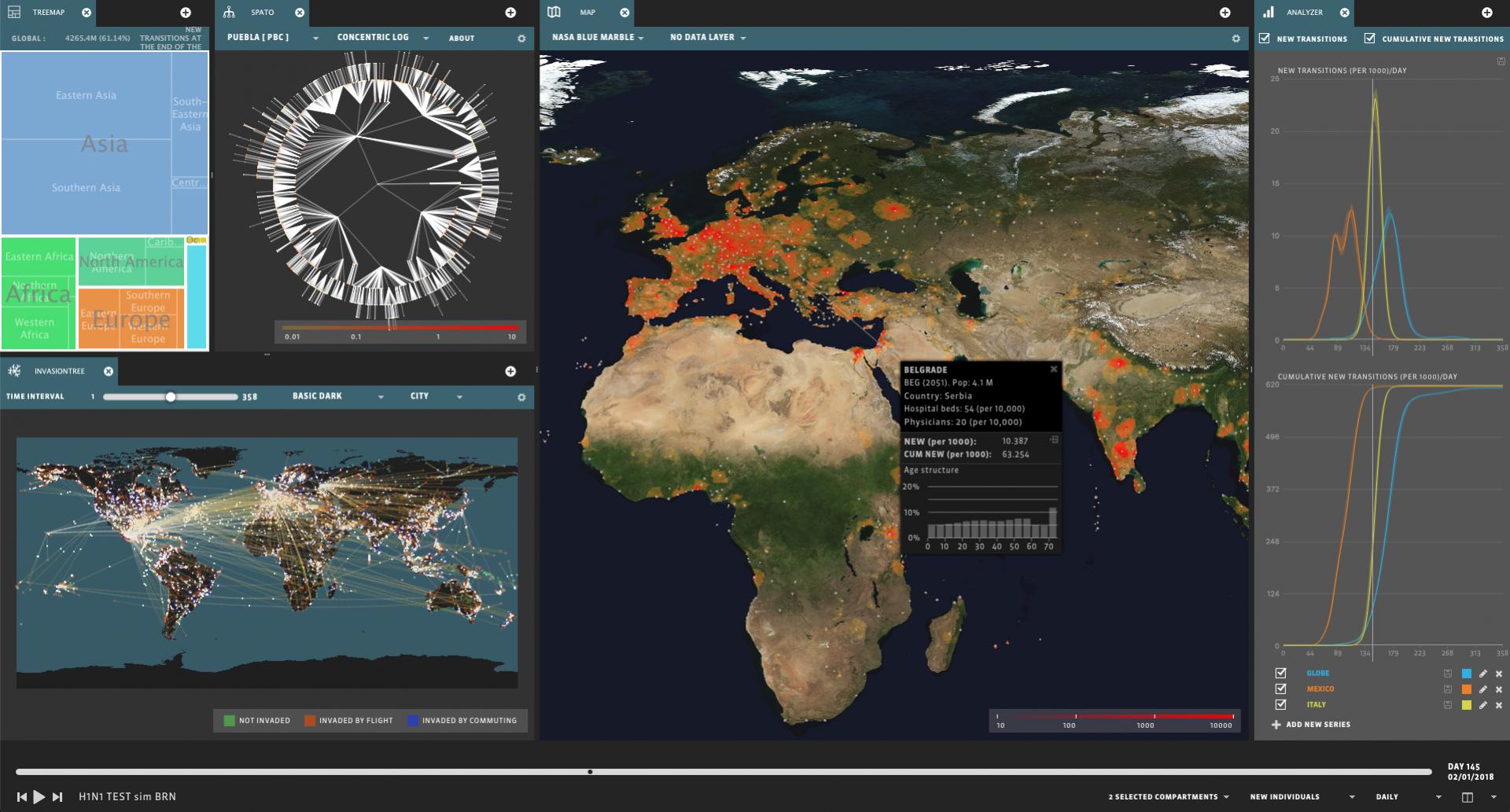
GLEAMviz-simulatorn är ett klient/server-program som implementerar den GLEAM-modell som är avsedd att användas av epidemiologer och policyansvariga för att analysera pandemiska hot och förutse deras utveckling samt att vara till hjälp för att ta itu med utmaningarna med att utveckla interventionsstrategier som minimerar pandemiers följder.
GLEAM är en stokastisk metapopulationsmodell som använder tre fundamentala skikt: befolkningsskiktet, mobilitetsskiktet och sjukdomsskiktet. Världsbefolkningen är uppdelad i mer än 3200 subpopulationer, centrerade kring de större flygknutpunkterna: befolkningsskiktet använder data från projektet Gridded Population of the World av SEDAC (https://sedac.ciesin.columbia.edu/data/collection/gpw-v4). Dessa folkräkningsområden är sammanlänkade via två olika mobilitetsnätverk; långdistansmobilitet använder data om flygpassagerare som samlats in från OAG, medan kortdistansmobilitet simuleras med pendlingsdata. I varje subpopulation använder sjukdomsdynamiken en kompartmentmodelleringsmetod: utvecklingen av epidemin bestäms av ekvationer som beskriver övergången av individer mellan olika befolkningsbestånd.
GLEAM-modellen har utvidgats på olika sätt, till exempel genom att den integrerats med detaljerade agentbaserade modeller på landsnivå, och har använts och validerats i många verklighetsbaserade scenarion, med början 2009 under H1N1-influensapandemin. Genom att använda GLEAMviz-simulatorn är det möjligt att utföra scenarioanalyser och uppskatta effektiviten hos olika begränsningsstrategier.
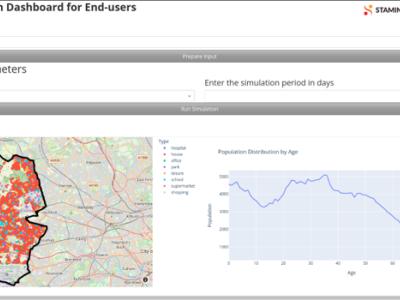
Med GLEAMviz-klienten kan användarna med hög flexibilitet designa simuleringar av utbrott av infektionssjukdomar. Det är möjligt att definiera komplexa biosektioneringar och lägga in övergångsparametrarna på ett strukturerat sätt. Därefter kan man, efter att begynnelsevillkoren angetts, simulera utvecklingen av en pandemi och uppskatta relevanta kvantiteter som incidens, utbrottstid osv. Verktyget gör det möjligt att simulera olika begränsningsstrategier, dvs. användning av läkemedel eller vacciner, och jämföra den förutsagda utvecklingen med referensscenariot.
Supported Use Cases
Modelling the outbreak of a new COVID-19 strain in the Valencia region
 |
Portfolio of Solutions web site has been initially developed in the scope of DRIVER+ project. Today, the service is managed by AIT Austrian Institute of Technology GmbH., for the benefit of the European Crisis Management. PoS is endorsed and supported by the Disaster Competence Network Austria (DCNA) as well as by the STAMINA and TeamAware H2020 projects. |